精准识别T细胞抗原是设计癌症mRNA疫苗最关键的一环。T细胞抗原经人类白细胞抗原(HLA)呈递后,能被T细胞表面受体(TCR)识别,激活T细胞增殖并释放细胞因子,进而靶向清除病原体,如细菌、病毒或肿瘤细胞。抗原与TCR的原子之间的接触(如氢键或盐桥)延长了HLA-抗原-TCR复合物的相互作用时间,是驱动T细胞免疫响应的关键因素。然而,如何揭示抗原与HLA、抗原与TCR的原子之间相互作用规律,进一步建立在原子水平识别T细胞抗原的人工智能模型,仍然面临着巨大挑战,是亟待解决的关键科学问题。
2025年6月4日,哈尔滨医科大学医工交叉学院蒋庆华、许召春、王平平、孙皓修研究团队在Nature Communications期刊发表题为“Identifying T cell antigen at the atomic level with graph convolutional network”的研究论文,报道了一种名为deepAntigen的人工智能模型,可以精准识别肿瘤新生抗原,并在肺癌、乳腺癌和胰腺癌上通过生物学实验验证了deepAntigen模型的准确性,为癌症治疗性mRNA疫苗设计奠定了基础。

基于序列的抗原与HLA/TCR相互作用预测方法忽略了原子级特征,导致其在识别能够激活免疫反应的抗原方面表现不佳并且难以解释突变对于免疫响应的影响。为了克服这一难题,deepAntigen将抗原、HLA和TCR的氨基酸序列转换为以原子为节点,化学键为边的图,将原子水平特征作为输入,可以精准识别能够被HLA呈递并被TCR识别的抗原。该方法实现了两大核心功能——deepAntigen_Seq与deepAntigen_Atom,前者预测抗原与HLA/TCR是否相互作用,后者预测抗原与HLA/TCR之间关键的原子间接触。由于HLA-抗原-TCR复合物晶体结构数据有限,导致原子间接触信息不足,该研究采用了大规模预训练思想和少样本学习策略解决这一问题。首先利用丰富的序列水平相互作用数据对deepAntigen进行预训练,图卷积神经网络隐式地学习关键的原子特征,然后利用少量的原子水平接触信息进行微调,有效地从序列水平相互作用数据中整合原子间接触的先验信息,提高了关键的原子间接触预测的准确性。
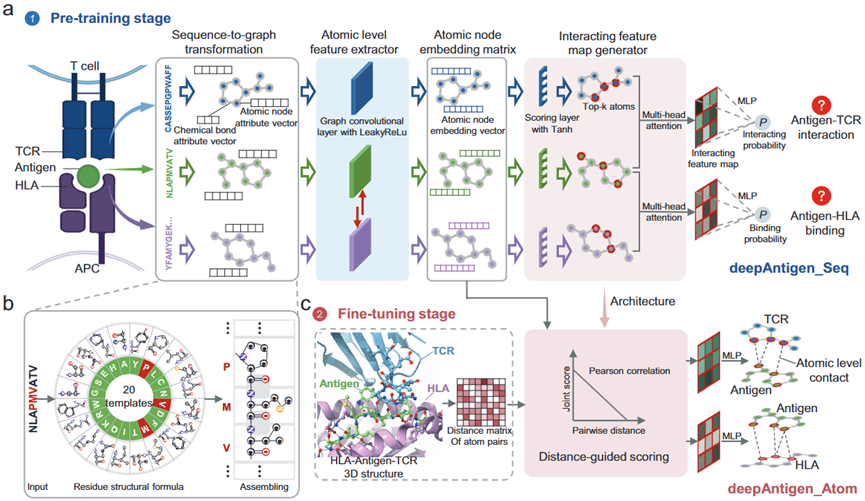
在原子水平预测HLA-Antigen-TCR相互作用的人工智能模型流程图
deepAntigen模型在多个任务和不同数据集中展示出卓越性能。在序列水平的抗原与HLA/TCR相互作用预测中,deepAntigen在准确性和泛化性等方面显著优于当前最先进的方法。在原子水平的接触预测方面,deepAntigen能够精准地识别关键的原子间接触,距离越近的原子对接触概率预测值越大,通过皮尔逊相关系数、ROC曲线下面积等指标进行综合评估,deepAntigen展示出优异的原子间接触预测性能。原子水平的接触预测功能使得deepAntigen能够精准识别抗原特异性TCR保守基序并捕获抗原单个残基突变对T细胞免疫响应程度的影响。
为了进一步验证deepAntigen识别癌症患者新抗原的有效性,该研究收集了5位癌症患者配对的癌症组织、癌旁组织和外周血样本,测定外显子组、转录组和免疫组库数据,将deepAntigen应用到每位患者的多组学测序数据上,每位患者筛选前20个候选新抗原,通过ELISPOT实验验证了筛选出的新生抗原能够激发强烈的T细胞免疫应答。
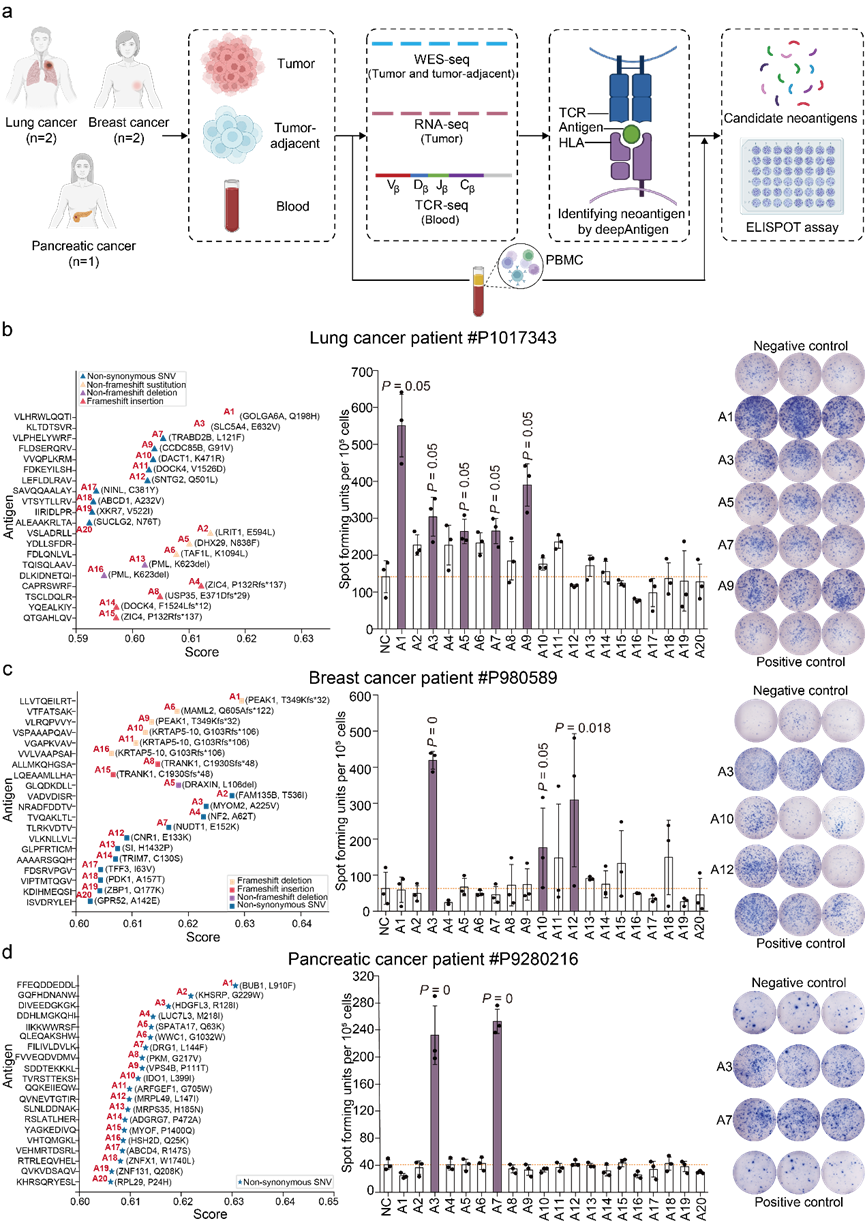
在肺癌、乳腺癌和胰腺癌上验证肿瘤新生抗原免疫原性
总而言之,deepAntigen人工智能模型能够在原子水平上精准识别肿瘤新生抗原,可为癌症mRNA疫苗设计提供技术支撑。
原文链接:https://doi.org/10.1038/s41467-025-60461-6
Que, J., Xue, G., Wang, T. et al. Identifying T cell antigen at the atomic level with graph convolutional network. Nature Communications 16, 5171 (2025).